Préliminaire
Ci-dessous, nous allons travailler avec une prédiction d’AlphaFold, avec la version de ColabFold. Cette prédiction peut prendre du temps, de 30 min à plusieurs heures selon la longueur de la protéine à prédire. De ce fait, il est recommandé de lancer le script de ColabFold en tâche de fond avant de réaliser la suite du TP.
Rendez-vous sur Uniprot et téléchargez la séquence de la protéine Q8J2T2.
Rendez-vous sur le notebook ColabFold: cliquez-ici.
Une fois sur le notebook, remplissez la séquence de la protéine (1) et donnez un nom à la session (2) (Voir figure ci-dessous).
Enfin, cliquez sur le menu Exécution > Tout exécuter (voir figure ci-dessous). Assurez-vous que le notebook s’exécute, puis continuez le TP. Vous reviendrez plus tard sur cette partie.
ATTENTION : NE FERMEZ PAS LA FENÊTRE, SINON LE CALCUL EST PERDU.
ATTENTION 2 : si le script fini, un popup apparaîtra pour vous proposer de télécharger les résultats. Soyez attentif ! Si vous le fermez par erreur, ne vous inquiétez pas, rendez vous en bas du notebook et lisez attentivement le guide.
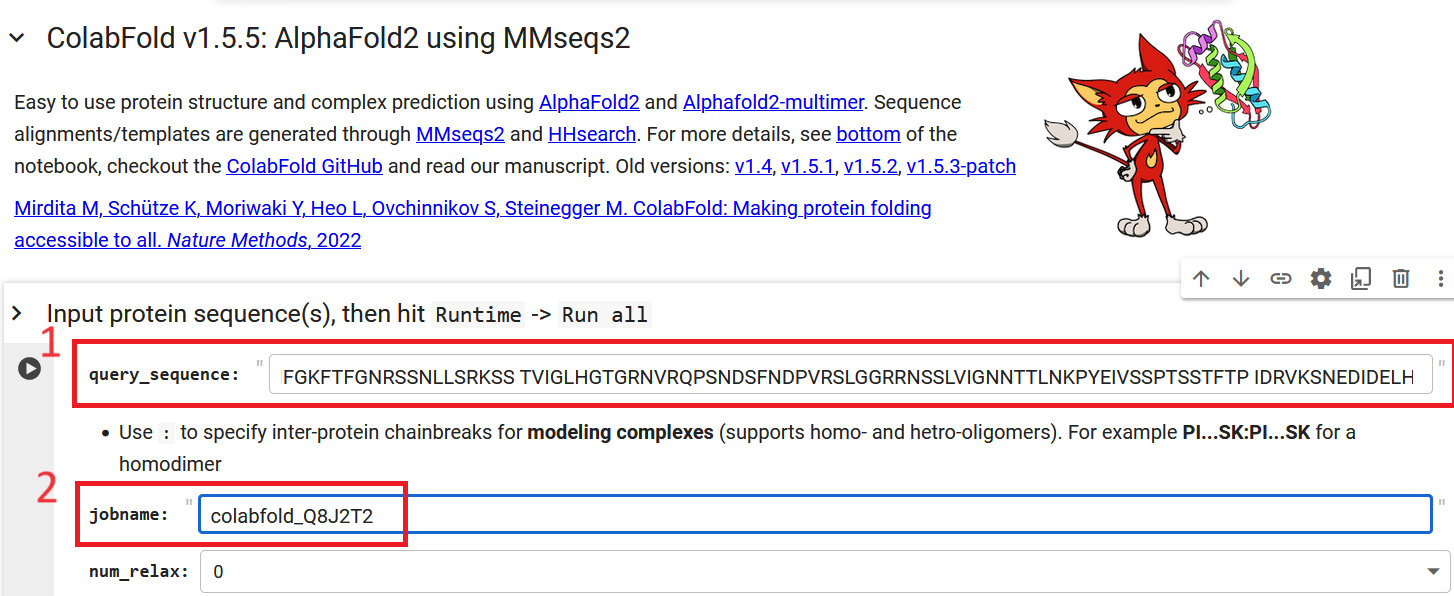
Figure: fournissez la séquence (1) et un nom pour récupérer les résultats (2)
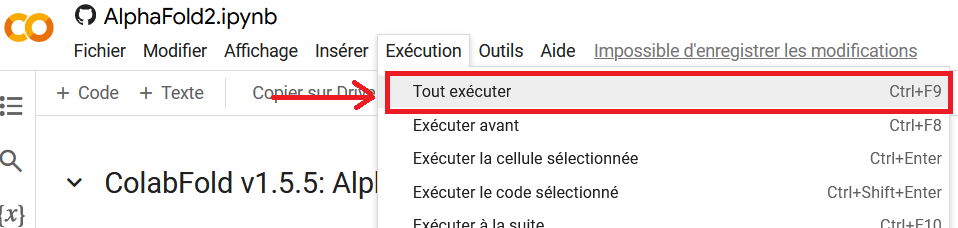
Figure: exécuter l’ensemble du script via le menu Exécution > Tout exécuter.
Obtenir la structure sur des protéines déjà prédites
De nombreux chercheurs ont réalisé des séquençages et déterminé la géométrie de nombreuses protéines. Ceux-ci peuvent être disponibles sur le projet AlphaFold Protein Structure Database. Pour explorer les résultats d’AlphaFold, nous prendrons dans un premier temps une protéine qui est connue.
Rendez vous sur Uniprot et chercher la protéine Q8J2T2. Vous trouverez déjà des informations concernant la structure de la protéine et les prédictions AlphaFold.
Pour obtenir l’ensemble des informations relatives aux prédictions d’AlphaFold, rendez-vous sur la base de données d’AlphaFold et chercher cette même protéine.
A l’aide des informations que vous avez obtenues, répondez aux questions suivantes en vous justifiant :
Analysez les données pLDDT. Est-ce que les régions de faibles confiances correspondent aux régions prédites comme désordonnées ?
Regardez le graphique des erreurs d’alignement prédites (PAE). Que signifie ce graphique ? Que comprenez et interprétez vous ?
Calculer la structure avec ColabFold
Dans les travaux ci-dessous, nous allons prédire la structure d’une protéine à l’aide d’AlphaFold. Souvenez-vous, pour des raisons pratiques vous avez déjà lancé le calcul.
Pour faciliter l’usage d’AlphaFold v2, des chercheurs mis à disposition un ensemble de code. Ces travaux se nomment ColabFold. La solution est basée du point de vue algorithmique sur AlphaFold v2 et utilise les outils de Google Colab. Pour les curieux, la proposition est décrite dans le papier suivant : ColabFold: making protein folding accessible to all.
Si vous ne l’avez pas encore fait, rendez-vous sur le notebook de ColabFold, entrez la séquence de la protéine et exécutez tout le notebook.
Si le script est terminé, un pop devrait vous avoir invité à télécharger les résultats. Si non, vous pouvez télécharger le “.zip” disponible dans le menu de gauche de Colab. Enfin, si le script n’est pas encore terminé, vous pouvez téléchargez les résultats intermédiaires “.pdb” disponible, eux aussi dans le menu de gauche de colab.
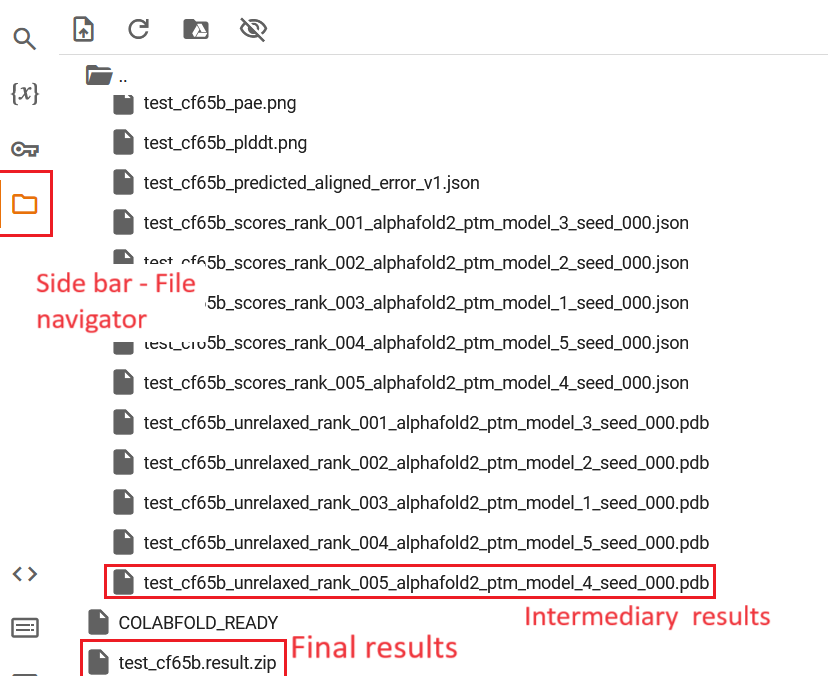
Figure: Navigateur de fichier - Sidebar Menu
Rendez-vous sur la base de donnée AlphaFold et téléchargez le fichier “.pdb” pour la protéine Q8J2T2.
Ouvrez les fichiers “.pdb” avec ChimeraX, ou tout autre logiciel similaire. Comparez. Quelles sont vos observations ?
(A faire à la maison) Appliquez cette séquence mais en réalisant une modification mineure de la protéine et évaluer son impact. Quelles sont vos observations ?
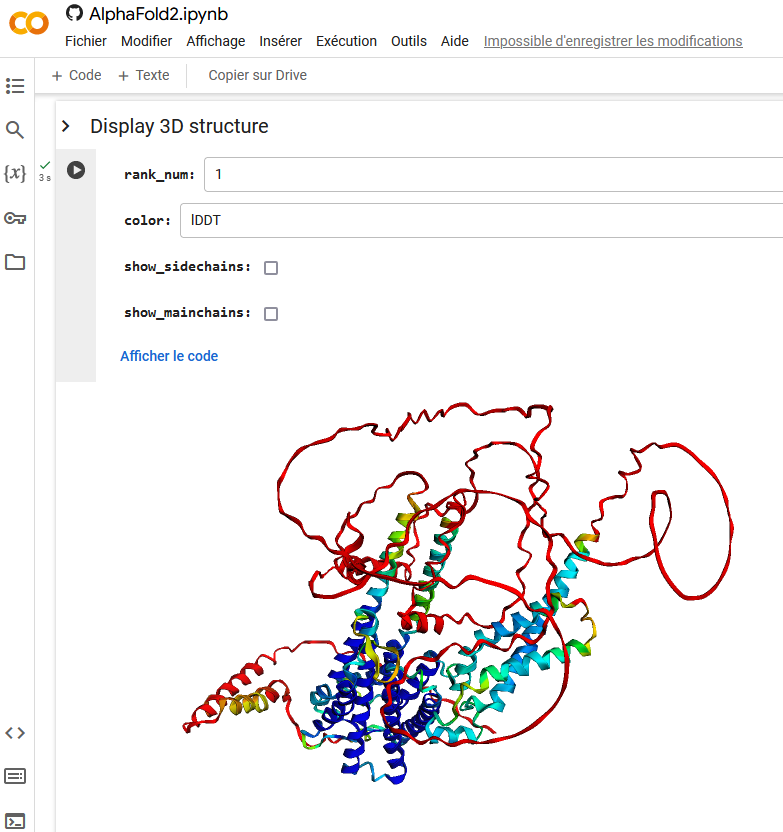
Figure: Exemple de résultat obtenu avec ColabFold